普段は自作の統計プログラムHADを使って分析していますが,人にいろいろ教えるときにはRも使っている清水です。
さて,今回はRで重回帰分析で交互作用を検討する方法について解説します。
昔,Rで重回帰分析で交互作用を検討するためのコードをアップしていたのですが,最近はもっと便利にできるパッケージがあるようです。というか僕が知らなかっただけか・・・
これについては,DARMという広大の院生・ポスドクが中心となってやっている勉強会のページで知りました(ここ)。まぁネタぱくってる,っていう話なんですけど(笑)。ぜひDARMのページも見てやってください。
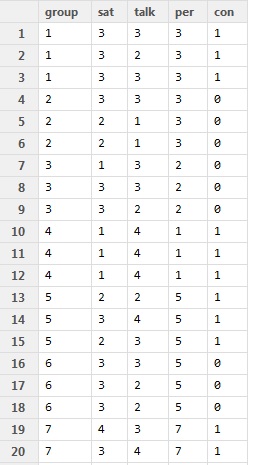
ここでは,次のようなサンプルデータを使います。20しか見えませんが,200人のデータです。サンプルデータはこちらからダウンロードできます。
これを”dat”に読み込みます。
dat <- read.csv("sampledata.csv")
まずは2要因の回帰分析を行ってみましょう。ここでは,従属変数はsat,独立変数はtalk,調整変数はperです。
で,使うパッケージはpequodです。そして,lmres関数を使います。
library(pequod)
model1 <- lmres(sat~talk*per, centered =c("talk", "per"), data =dat)
summary(mode1)
sat ~ talk*perというモデル式は,talkとperの主効果と,talkとperの交互作用を投入する,という意味です。つまり,sat~talk+per+talk:perと同じ意味です。
次に,centered=では,中心化する(平均を0にする)変数を指定します。変数名を""でくくるのを忘れずに。基本的に,回帰分析の交互作用を検討する場合,平均を0にします。それは,多重共線性を避けるためです。
結果は次のようになります。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 |
Models R R^2 Adj. R^2 F df1 df2 p.value Model 0.467 0.218 0.211 27.576 3.000 296 9.4e-16 *** --- Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1 Residuals Min. 1st Qu. Median Mean 3rd Qu. Max. -2.3160 -0.4539 -0.0764 0.0000 0.6840 2.2810 Coefficients Estimate StdErr t.value beta p.value (Intercept) 3.41647 0.05116 66.77491 <2e-16 *** talk 0.26572 0.05192 5.11783 0.2649 <2e-16 *** per 0.14054 0.02941 4.77880 0.2482 <2e-16 *** talk.XX.per 0.13022 0.03035 4.29012 0.2234 2e-05 *** --- Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1 Collinearity VIF Tolerance talk 1.0148 0.9854 per 1.0216 0.9789 talk.XX.per 1.0270 0.9737 |
talk xx perの項が有意であることに注目しましょう。交互作用項が有意だったので,単純主効果を検討しましょう。
単純主効果は,以下のコードで検討します。
model2 <- simpleSlope(model1, pred="talk", mod1 = "per")
summary(model2)
simpleSlopeの二つ目のSが大文字なのに注意。predは予測変数,つまり独立変数を指定します。つまり,talkがそれになります。mod1は,調整変数の一つ目,ということで,perを指定します。結果は以下のようになります。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 |
** Estimated points of sat ** Low talk (-1 SD) High talk (+1 SD) Low per (-1 SD) 3.1329 3.2064 High per (+1 SD) 3.1731 4.1534 ** Simple Slopes analysis ( df= 296 ) ** simple slope standard error t-value p.value Low per (-1 SD) 0.0370 0.0779 0.48 0.63 High per (+1 SD) 0.4944 0.0708 6.99 <2e-16 *** --- Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1 ** Bauer & Curran 95% CI ** lower CI upper CI per -4.1656 -1.078 |
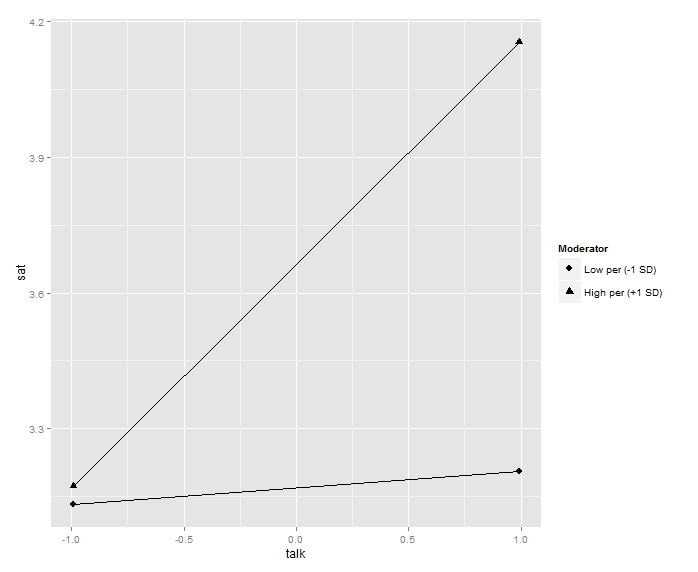
perが高群(+1SD)の場合に,talkの効果は有意ですが,低群(-1SD)の場合,非有意であることがわかります。
次に,単純効果をグラフ表示したい場合には,PlotSlope関数を使います。
PlotSlope(model2)
大文字,小文字の違いに注意。これを走らせると,以下のようなグラフが表示されます。
なかなかいい感じですね。
なお,このlmres関数では,交互作用項が二つ以上でも単純主効果の分析ができます。たとえば,下のように3要因の重回帰分析を実行した場合,
model1 <- lmres(sat~talk*per*con,centered=c("talk","per","con"),data=dat)
結果は以下のようになります。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 |
Models R R^2 Adj. R^2 F df1 df2 p.value Model 0.497 0.247 0.229 13.682 7.000 292 2.6e-15 *** --- Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1 Residuals Min. 1st Qu. Median Mean 3rd Qu. Max. -2.3060 -0.4832 -0.0182 0.0000 0.6554 2.1850 Coefficients Estimate StdErr t.value beta p.value (Intercept) 3.40850 0.05648 60.35227 < 2e-16 *** talk 0.25540 0.05841 4.37218 0.2546 2e-05 *** per 0.12658 0.03086 4.10147 0.2236 5e-05 *** con 0.19472 0.11295 1.72393 0.0981 0.08578 . talk.XX.per 0.12690 0.03268 3.88297 0.2177 0.00013 *** talk.XX.con 0.03388 0.11683 0.29000 0.0156 0.77202 per.XX.con 0.00480 0.06173 0.07771 0.0042 0.93811 talk.XX.per.XX.con 0.15150 0.06536 2.31791 0.1303 0.02115 * --- Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1 Collinearity VIF Tolerance talk 1.3152 0.7603 per 1.1520 0.8680 con 1.2550 0.7968 talk.XX.per 1.2189 0.8204 talk.XX.con 1.1213 0.8918 per.XX.con 1.1412 0.8763 talk.XX.per.XX.con 1.2256 0.8160 |
単純効果の検定も,2要因の時と同様に,
model2 <- simpleSlope(model1,pred="talk",mod1 = "per",mod2="con")
と書きます。ご丁寧に,ボンフェローニの修正p値も出力します。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 |
** Estimated points of sat ** Low talk (-1 SD) High talk (+1 SD) Low per (-1 SD) , Low con (-1 SD) [1] 2.9453 3.2404 Low per (-1 SD) , High con (+1 SD) [2] 3.3625 3.1965 High per (+1 SD) , Low con (-1 SD) [3] 3.2038 3.8543 High per (+1 SD) , High con (+1 SD) [4] 3.1095 4.3556 ** Simple Slopes analysis ( df= 292 ) ** simple slope standard error t-value p.value Low per (-1 SD) , Low con (-1 SD) [1] 0.1488 0.1399 1.06 0.2884 Low per (-1 SD) , High con (+1 SD) [2] -0.0837 0.1007 -0.83 0.4063 High per (+1 SD) , Low con (-1 SD) [3] 0.3280 0.0938 3.50 0.0005 *** High per (+1 SD) , High con (+1 SD) [4] 0.6285 0.1234 5.09 <2e-16 *** --- Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1 ** Slope Difference Test (( df= 292 ); Dawson & Richter, 2006) ** t-value p.value High per (+1 SD) , High con (+1 SD) [4] vs. High per (+1 SD) , Low con (-1 SD) [3] 1.9371 0.0537 High per (+1 SD) , High con (+1 SD) [4] vs. Low per (-1 SD) , High con (+1 SD) [2] 4.5459 0.0000 High per (+1 SD) , High con (+1 SD) [4] vs. Low per (-1 SD) , Low con (-1 SD) [1] 2.5712 0.0106 High per (+1 SD) , Low con (-1 SD) [3] vs. Low per (-1 SD) , Low con (-1 SD) [1] 1.1438 0.2536 High per (+1 SD) , Low con (-1 SD) [3] vs. Low per (-1 SD) , High con (+1 SD) [2] 2.9919 0.0030 Low per (-1 SD) , High con (+1 SD) [2] vs. Low per (-1 SD) , Low con (-1 SD) [1] -1.4995 0.1348 Bonferroni.p High per (+1 SD) , High con (+1 SD) [4] vs. High per (+1 SD) , Low con (-1 SD) [3] 0.3222 High per (+1 SD) , High con (+1 SD) [4] vs. Low per (-1 SD) , High con (+1 SD) [2] <2e-16 *** High per (+1 SD) , High con (+1 SD) [4] vs. Low per (-1 SD) , Low con (-1 SD) [1] 0.0638 . High per (+1 SD) , Low con (-1 SD) [3] vs. Low per (-1 SD) , Low con (-1 SD) [1] 1.0000 High per (+1 SD) , Low con (-1 SD) [3] vs. Low per (-1 SD) , High con (+1 SD) [2] 0.0181 * Low per (-1 SD) , High con (+1 SD) [2] vs. Low per (-1 SD) , Low con (-1 SD) [1] 0.8090 --- Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1 |
また,出力したモデルのF_changeという要素を見れば,階層的重回帰分析の要領で,交互作用ごとのΔR2乗値が確認できます。
|
1 2 3 4 5 6 7 8 9 10 11 |
model1$F_change Analysis of Variance Table Model 1: sat ~ talk + per + con Model 2: sat ~ talk + per + con + talk.XX.per + talk.XX.con + per.XX.con Model 3: sat ~ talk + per + con + talk.XX.per + talk.XX.con + per.XX.con + talk.XX.per.XX.con Res.Df RSS Df Sum of Sq F Pr(>F) 1 296 241.56 2 293 226.74 3 14.8214 6.4796 0.0002935 *** 3 292 222.64 1 4.0965 5.3727 0.0211455 * |
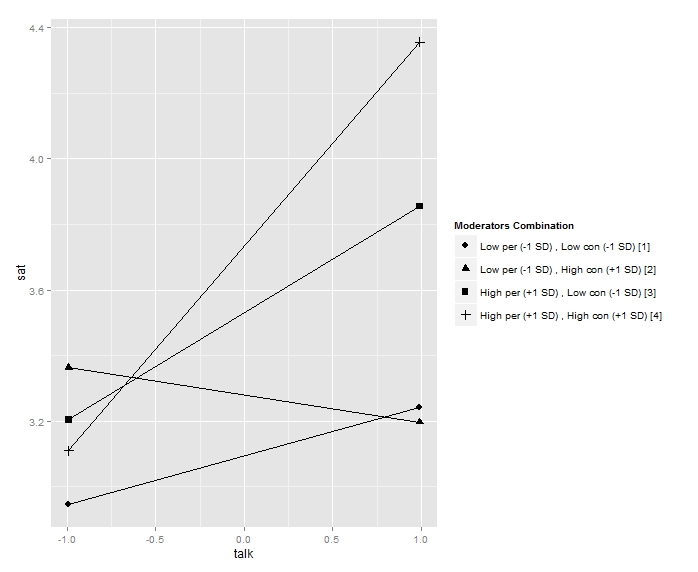
グラフは下のようになりました。
すべての群が同じグラフに表示されるのは,便利なのか見にくいのかは評価が分かれるところですが,Rでここまで簡単に重回帰の交互作用を検討できるパッケージがあるのはとても便利ですね。